Optische Keimdetektion
Schnelle Verfahren zur Erkennung von Keimbelastungen werden zunehmend wichtiger. Nicht zuletzt aufgrund der immer engeren international und global verstrickten Produktionsketten und höheren Automatisierungsgraden können Kontaminationen sich sehr schnell sehr weit verbreiten.
Beim Stichwort Keimdetektion sind die erste Assoziation oft Krankenhäuser, denn natürlich muss dort streng auf Hygiene geachtet und die Einhaltung der Vorgaben auch kontrolliert werden.
Systemische Präventionsansätze (HACCP, GMP u.v.m.) sind jedoch auch in vielen anderen Industrien fester Bestandteil des Arbeitsalltags und immer häufiger auch durch Normen vorgeschrieben. Eine große Rolle spielen sie z.B. in der Arzneimittel- und auch in der Lebensmittelproduktion.
Das Ziel der präventiven Ansätze ist es immer, die Produktionsabläufe selbst schon durch bestimmte Prozesse so abzusichern, das Infektionen oder Kontaminationen mit Schadstoffen oder Toxinen verhindert werden, bevor Menschen erkranken, verunreinigte Lebensmittel ausgeliefert oder Produkt-Rückrufe gestartet werden, um den Schaden einzudämmen.
Eine zentrale Rolle spielen dabei Hygieneprozesse, etwa bei der Werkzeug- und der Oberflächenreinigung.
Woher weiß man nun aber, ob die Reinigung einer Oberfläche erfolgreich alle Keime abgetötet hat? Hier spielen ja viele Faktoren eine Rolle: Art und Menge des Reinigungsmittels, die Art der Oberfläche, die Durchführung der Reinigung selbst, die Häufigkeit der Reinigung usw. usf. Vielleicht macht man sich auch viel zu viel Arbeit, und eine Oberfläche ist bereits nach der Hälfte des Vorgangs steril.
Das Ergebnis der Reinigung muss also geprüft werden. Das ist die Aufgabe von Keimdetektionsverfahren.
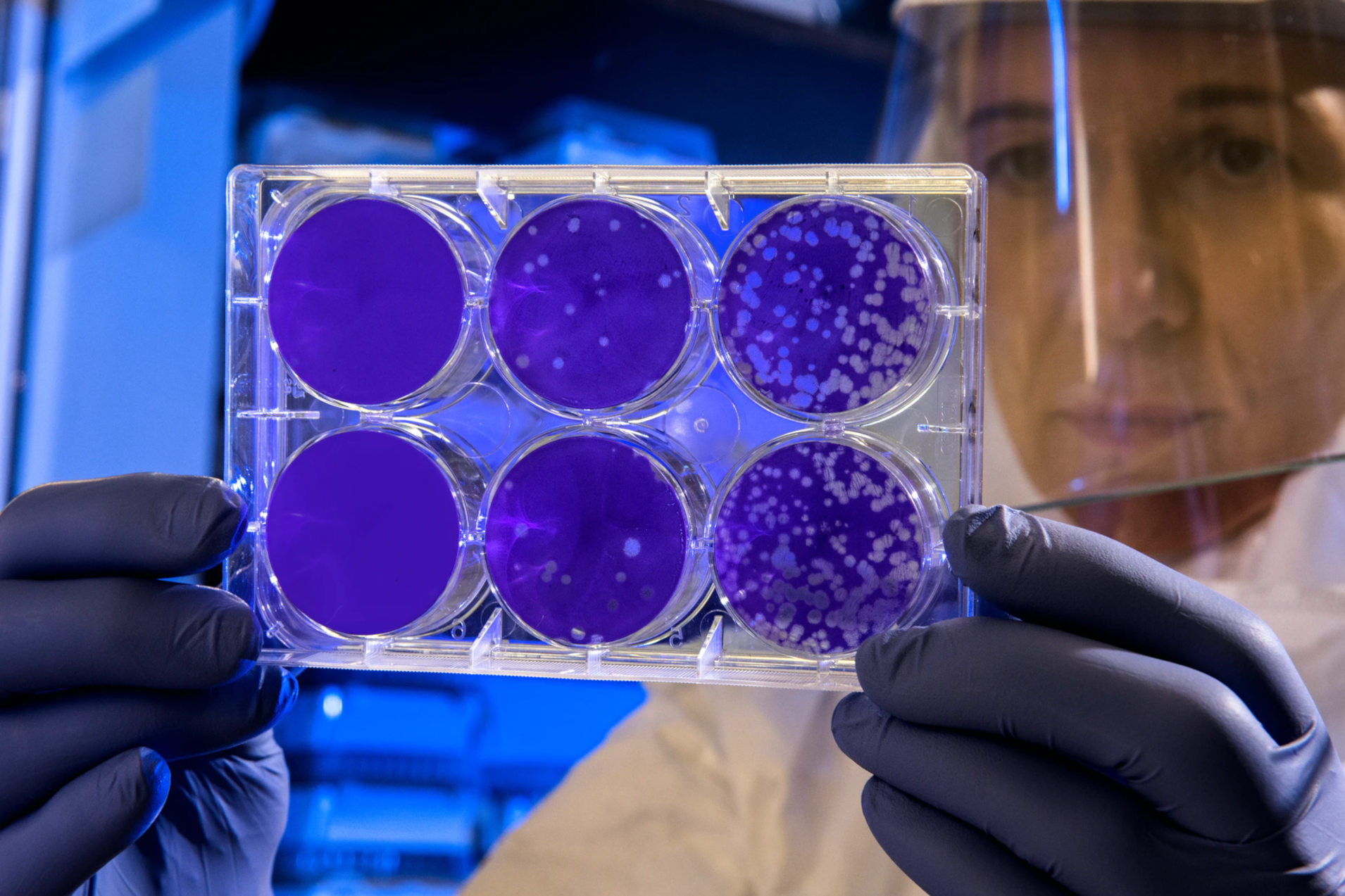
Klassisch werden in diesem Fall Proben von der Oberfläche genommen, auf Petrischalen mit Nährlösung übertragen und einige Tage lang bebrütet, d.h. in einer möglichst keimfreundlichen Umgebung zum Wachstum angeregt. Nach einigen Tagen und vielen Vermehrungszyklen haben sich die Keime dann so stark vermehrt, dass sie als unterschiedliche Flecken auf der Nährlösung für das bloße Auge sichtbar werden.
Diese Flecken, die koloniebildenden Einheiten (KBE), werden dann ausgezählt.
Dieses Verfahren ist robust und bewährt, aber es ist auch sehr langsam. Darum wurden verschiedene Schnellverfahren zur Keimerkennung entwickelt, die mindestens gleichwertige Ergebnisse in deutlich kürzerer Zeit liefern.
ATP Biolumineszenz
Eines der häufigsten Schnellverfahren macht sich dieselbe chemische Reaktion zunutze, die auch Glühwürmchen zum Glühen bringt.
Bei einem ATP-Schnelltest wird mithilfe eines Tupfers eine Probe von der zu prüfenden Oberfläche genommen und dann in eine Küvette (ähnlich einem Reagenzglas) gesteckt. Wenn Keime in der Probe enthalten waren, dann befindet sich jetzt Adenosintriphosphat (ATP) im Behälter, denn ATP ist als Energieträger in den allermeisten Zellen enthalten.
In die Küvette wird zusätzlich ein Gemisch aus Luciferase und Luciferin gefüllt. Zusammen mit dem ATP kommt es zur Luciferase-Luciferin-Reaktion, bei der das ATP abgebaut wird. Pro ATP-Molekül wird dabei jeweils überschüssige Energie freigesetzt, die in Form eines Lichtquants an die Umwelt abgegeben wird.
Das ist die Quelle des Leuchtens bzw. der Lumineszenz. Je mehr Zellen, d.h. ATP in der Probe enthalten war, desto stärker wird das Leuchten ausfallen.
Das Ergebnis ist trotzdem recht leuchtschwach, und vor allem können wir mit bloßem Auge die relativen Intensitätsunterschiede nicht genau genug unterscheiden. Die Auswertung der Biolumineszenz erfolgt deshalb meist mithilfe von Maschinen.
Da unterschiedliche Keimtypen unterschiedlich starkes Leuchten produzieren, ist es leider nicht ohne weiteres möglich, die Leuchtstärke eindeutig in die Anzahl koloniebildender Einheiten umzurechnen. Es ist aber grundsätzlich möglich, wenn es nur ein bestimmter Keim sein kann, dessen ATP-Biolumineszenz zuvor ausgemessen wurde.
ATP-Schnelltests sind einfach einsetzbar, verhältnismäßig günstig und die mobilen Geräte selten größer als eine Packung Kaffee, darum ist diese Testform sehr weit verbreitet.
DEFT (Direct Epifluorescent Filter Technique)
Auch die DEFT-Methode funktioniert optisch, nutzt jedoch Fluoreszenz, nicht Lumineszenz.
Es wird wieder eine Probe entnommen, mit einer Lösung verdünnt und anschließend durch eine spezielle Membran filtriert. Die Lösung enthält zusätzlich zwei verschiedene Fluoreszenzmarker, spezielle Farbstoffe, die sich nur an ganz bestimmte Teile einer Zelle anheften und bei einer ganz bestimmten Wellenlänge leuchten, wenn sie durch anderes Licht angeregt werden.
DEFT nutzt nun einmal einen Fluoreszenzmarker, um Esterase zu markieren, einem Stoff, der eine wichtige Rolle bei der Stoffwechselaktivität einer Zelle spielt. Ein aktiver Stoffwechsel heißt, dass diese Zelle lebt. Dieser Farbstoff markiert also lebende Zellen.
Ein zweiter Farbstoff verbindet sich mit DNA. Diese ist bekanntermaßen normalerweise geschützt im Zellkern. Um die DNA zu erreichen, muss der Zellkern also zugänglich sein, d.h. diese Zelle ist tot.
Bestrahlt man nun die Probe mit genau dem Licht, dass diese beiden Farbstoffe zum Fluoreszieren bringt, dann können die koloniebildenden Einheiten anschließend mithilfe eines Mikroskops ausgezählt werden. Ursprünglich geschah das manuell, heutzutage gibt es immer mehr Geräte, bei denen die Auszählung von einer Software übernommen wird.
Die Probenaufbereitung ist hier verhältnismäßig aufwändig und die Laborgeräte nicht gerade günstig, darum wird DEFT hauptsächlich für bestimmte Nischen eingesetzt, in denen die sehr hohe Präzision dieser Technik besonders vorteilhaft ist.
Durchflusszytometrie bzw. Festphasenzytometrie
Die Durchflusszytometrie bedient sich der Tatsache, dass viele Zellbestandteile eine ganz bestimmte Autofluoreszenz aufweisen, wenn man sie mit bestimmten Lichtfrequenzen bestrahlt, d.h. sie weisen von sich aus Fluoreszenzeigenschaften auf, ohne Zugabe eines speziellen Farbstoffs.
Die Keimerkennung mithilfe der Durchflusszytometrie beginnt ebenfalls mit der Entnahme einer Probe. Auch hier wird der Tupfer zunächst mit einer Lösung behandelt, um die Keime vom Tupfer in die Flüssigkeit zu bekommen.
Diese Flüssigkeit wird nun durch einen sehr engen Kanal geschickt, so dass praktisch nur einzelne Zellen hintereinander die engste Stelle passieren können.
An diesem Punkt werden nun mit mehreren Laserstrahlen ganz bestimmter Wellenlängen aus verschiedenen Richtungen bestrahlt und das Ergebnis jeweils mithilfe von Photodioden gemessen. Das Ganze passiert recht schnell, es können problemlos mehr als 1000 Zellen in der Sekunde geprüft werden.
Aufgrund der charakteristischen Fluoreszenz-Antworten und -intensitäten können auf diese Weise viele verschiedene Zellen nicht nur gezählt, sondern sogar identifiziert werden.
Die Durchflusszytometrie wird am häufigsten für Proben verwendet, die ohnehin schon Flüssigkeiten sind, beispielsweise bei Trinkwasser oder Blutproben. Hier kann auch zusätzlich mit Fluoreszenzfarbstoffen gearbeitet werden, um ähnlich wie bei DEFT tote Zellen getrennt auszählen zu können.
PCR (Polymerase-Kettenreaktion)
Zwar nur im letzten Schritt optisch, aber aufgrund der Corona-Pandemie eine heutzutage häufig genannte Methode ist der PCR-Test.
Bei PCR-Tests werden genetische Informationen direkt detektiert. Dafür wird das genetische Material in einer Probe zunächst vervielfältigt. In der lebenden Zelle liegt die DNA in der bekannten Doppelhelix-Form vor. Im Falle einer natürlichen Zellteilung wird diese doppelte Kette chemisch aufgetrennt und jeweils ein neues Gegenstück hergestellt, so dass am Ende des Vorgangs zwei identische Ketten vorliegen.
Das PCR-Verfahren imitiert diese Vorgänge im Labor. Die Auftrennung erfolgt hier jedoch mit Hilfe hoher Temperaturen: Erhitzt man die DNA-Kette auf ca. 95 °C, dann spaltet sie sich von allein auf.
Der Probe wird zusätzlich eine Polymerase hinzugefügt, die dazu führt, dass sich die DNA replizieren kann. Die dafür nötigen DNA-Bausteine, die Desoxyribonukleosidtriphosphate, werden ebenfalls hinzugegeben.
Es kann auf diese Weise derzeit noch nicht die gesamte DNA repliziert werden, sondern nur bestimmte Abschnitte. Das ist im Falle der Keimerkennung jedoch kein Nachteil, denn meist ist man hier an recht kurzen Abschnitten interessiert. Es gibt beispielsweise einen bestimmten kurzen Abschnitt, den alle Bakterien enthalten, und der deshalb häufig bei der Keimdetektion Verwendung findet.
Anfang und Ende des gewünschten Abschnitts werden mit sogenannten Primern markiert, die ebenfalls beigemischt werden. Im ersten Zyklus nach der Auftrennung bindet der eine Primer, im nächsten auch der zweite, so dass erst zu diesem Zeitpunkt das PCR-Produkt in korrekter Länge vorliegt.
Die Zyklen von Erhitzung und anschließender Vervielfältigung führen so zur Verdopplung des interessanten Abschnitts. Je nach ursprünglich vorhandener Konzentration wird er deshalb viele Male wiederholt.
Bei einer kontaminierten Probe würden nun ausreichend viele PCR-Produkte vorliegen, um einen sicheren Nachweis zu ermöglichen.
Dazu wird die Probe in ein besonderes, elektrisch leitfähiges Gel eingebracht. Durch Anlegen einer Spannung sortieren sich die DNA-Stränge entsprechend ihrer Länge. Als Maßstab wird gleichzeitig eine sogenannte DNA-Leiter hinzugefügt, die verschiedene Stränge bekannter Länge enthält und damit einen direkten Vergleich der Probe mit den Referenzlängen ermöglicht. So weit, so gut, nur muss das Material nun auch noch sichtbar gemacht werden. Auch hier kommen wieder Fluoreszenzmarker ins Spiel, die an DNA binden. Diese werden mit der entsprechenden Wellenlänge beleuchtet und strahlen dann bei einer anderen, bekannten Wellenlänge, die meist im sichtbaren Bereich liegt, aber auch im langwelligen UV-Bereich verortet sein kann.
PCR-Tests können bereits sehr geringe Keimbelastungen erkennen, dauern jedoch mehrere Stunden und erfordern die entsprechende Labor-Ausstattung.
Ausblick
Die aufgezeigten Schnellerkennungsmethoden können teilweise bereits sehr kleine Mengen koloniebildender Einheiten nachweisen - jedoch wird die Präzision durch eher längere Testzeiträume und höhere Kosten erkauft. Damit eignen sie sich sehr gut für stichprobenhafte Kontrollen. Für kontinuierliche Messungen, die regelmäßig als Element eines HACCP- oder GMP-Prozesses zur Bioburden-Bestimmung durchgeführt werden können, wären zusätzlich Techniken wünschenswert, die direkt im Feld innerhalb weniger Minuten ausreichend präzise Ergebnisse liefern könnten.



